Thème "Ingénierie Moléculaire"
Quelques mots sur le thème
Une expertise dans l'ingénierie des protéines membranaires d’oxydoréduction
L’équipe I2M est également reconnue pour son savoir-faire dans le domaine de l’ingénierie des protéines, en particulier les cytochromes P450 et leurs donneurs d’électrons. Nous avons développé des approches originales de création de banques combinatoires et de cartographie fonctionnelle des enzymes modifiés. Les méthodes d’ingénierie que nous utilisons visent soit des enzymes individuelles (modification des activités des enzymes naturelles, ingénierie des sites actifs ou de reconnaissance) soit des assemblages macromoléculaires (formation de complexes rédox, formation de la coque des microcompartiments bactériens, assemblages d’enzymes individuels). L’ingénierie moléculaire est parfaitement intégrée au thème ingénierie métabolique car les deux poursuivent le même objectif : obtenir des systèmes métaboliques synthétiques efficaces. Les modèles enzymatiques utilisés sont des oxydases (cytochromes P450), des terpènes synthétases et les enzymes de la voie de production des caroténoïdes (désaturases, cyclases, dioxygénases, etc.).
Les projets
Projet
Bacterial Microcompartments
La synthèse continue exige le développement de nouveaux outils biotechnologiques pour améliorer l’efficacité des cascades métaboliques créées dans des micro-organismes donnés. Deux stratégies majeures pour atteindre cet objectif sont la colocalisation spatiale des enzymes conçues et l’optimisation de leur orientation relative. Dans ce sens, notre première intention est d’apprendre de la nature, en contribuant à notre compréhension de la fonction des micro-compartiments bactériens (BMC). Les BMC sont des assemblages macromoléculaires qui encapsulent naturellement des enzymes qui participent à divers processus métaboliques. Nous étudions par divers moyens biophysiques le mécanisme qui conduit à l’assemblage de la coquille des BMC. L’objectif du second axe de ce projet est d’établir la composition précise des unités d’assemblage naturelles pour ensuite concevoir de nouvelles plateformes hexamériques.
Projet
Combinatoire des séquences linkers sur l’activité des protéines fusions artificielles.
Projet
Dynamique entre domaines au sein d’une oxidoréductase multidomaine
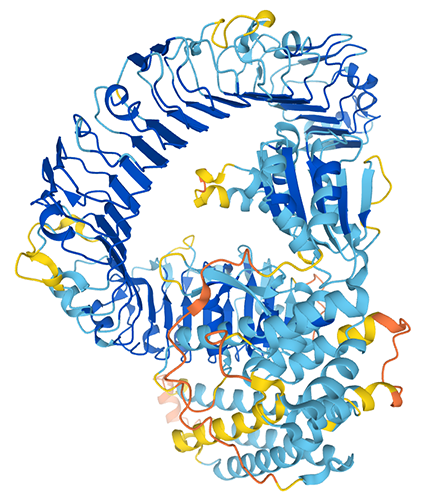
Dans l’organisme, les médicaments subissent un métabolisme spécifique qui s’effectue principalement par les enzymes P450. Une seule flavoenzyme, la NADPH-cytochrome P450 réductase (CPR), donne les électrons nécessaires aux P450s pour leur catalyse. Jusqu’à présent, la plupart des variations interindividuelles de ce métabolisme ont été étudiées sur les polymorphismes du P450. Les modifications de l’équilibre conformationnel de la CPR, causées soit par des mutations, soit par l’interaction avec de petites molécules, peuvent augmenter son affinité pour un P450 spécifique, au détriment des autres. L’objectif de ce projet est de déchiffrer les facteurs (membrane du reticulum endoplasmique, état d’oxydation et variantes polymorphes de la CPR) qui contrôlent les modifications structurales et la dynamique de la CPR ancrée dans la membrane et leur implication dans le métabolisme des médicaments et les déficiences fonctionnelles potentielles. Ce projet vise à fournir de nouveaux outils pour décrypter les facteurs moléculaires modulant le paysage dynamique de la CPR et la spécificité des interactions dans les complexes CPR/P450.
Projet AlphaFold2
AlphaFold et complexes protéiques
AlphaFold2, an AI deep-learning system developed by DeepMind surpasses in accuracy all previous computing attempts for protein structure predictions. Recent versions were successfully extended to modeling of protein complexes including in some instances the potential to address large scale conformational changes involved in catalysis. DeepMind and EMBL’s European Bioinformatics Institute (EMBL-EBI) have partnered to create AlphaFold Protein Structure DataBase (ADB) that covers the complete human and 47 other key organisms (mouse, yeast, Arabidopsis, etc.), as well as the manually curated UniProt database. As of April 2022, AlphaFold DB provides open access to 992,316 protein 3D structure entries, most of them with no experimental structure available. We are using AlphaFold2_advanced version to look at the structures of complexes between P450 enzymes and P450-associated redox proteins and to their alternative geometries.



